リカレンスプロットに距離補正を加えることで染色体の構造推定における空間的解像度が向上
Advantages
- リカレンスプロットに距離の補正を入れることで1細胞Hi-Cデータからの染色体の立体構造をより高精度に再構成することが可能
- 追加の工程は不要であり、既存の1細胞Hi-Cワークフローにスムーズに組み込める
Background and Technology
染色体の立体構造は、DNAやタンパク質間の物理的な相互作用を理解するうえで重要であり、エピゲノムや遺伝子発現に大きく関与する。こうした構造の解析には、従来Hi-C(genome-wide chromatin conformation capture)法が広く利用されてきた。Hi-Cは、核内で空間的に近接するDNA領域をクロスリンクし、制限酵素で切断・ライゲーションを経て生成されたDNA断片をシーケンスすることで、染色体の三次元構造を推定する手法である。1細胞Hi-Cは、Hi-Cを1細胞ごとに得る方法である。一般的な1細胞Hi-C解析では、まず低解像度のモデルを構築し、それをもとに高解像度の構造を推定する。しかし初期段階で得られる低解像度モデルの精度が、最終的な高解像度モデルの品質を左右するため、より高精度な低解像度再構成手法が求められていた。
筑波大学の平田教授らは、時系列解析で用いられるリカレンスプロット(Recurrence Plot, RP)を1細胞Hi-Cデータ解析に応用することで、染色体構造の再構成精度を高める新しい手法を開発した。リカレンスプロットは、時系列データのパターンや距離情報を可視化する数学的手法であり、距離行列に基づく構造復元に優れている。本手法では、局所的な距離に補正を入れることで、従来手法と比べて再構成精度が向上し、より高精度な染色体の三次元再構造の推定が可能となった。本手法は追加の実験工程を必要とせず、既存の1細胞Hi-Cデータを用いた計算処理のみで実行可能であるため、既存の解析に無理なく導入できる。これにより、TAD(Topologically Associating Domains)などの重要なドメイン構造の検出や、生物学的機能の解明への貢献が期待される。
Data
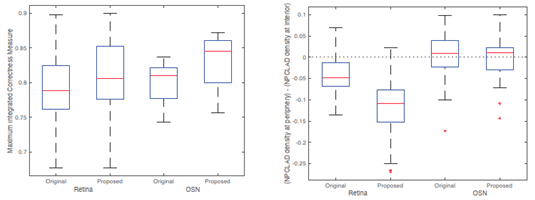
- Retina細胞とOSN細胞の公開されている1細胞Hi-Cデータを用いて、従来方法(Original)と本手法(Proposed)による構造再構成の精度指標(Integrated Correctness Measure)を比較したところ、いずれの細胞においても本手法が高い再構成精度を示すことが確認された。
- Lamina Associated Domain(LAD)の空間分布:Retina細胞およびOSN細胞におけるLAD分布を、NPC細胞を基準に評価した。OSN細胞ではOriginal・Proposedともに外側優位の分布を示す一方、Retina細胞ではProposed手法が内側移動傾向をより強く再現し、生物学的知見との整合性が確認された。
Expectations
筑波大学ではより緻密な染色体の三次元構造を推定する手法に関心のある受託解析・検査サービスを行っている企業様を探しています。技術評価やライセンス導入に向けた研究者との面談が可能です。ライセンス導入に向けた技術評価として、企業様がお持ちの1細胞Hi-Cデータを用いた本プログラムの評価をいただくことも可能です。
Patents
WO/2023/048251
Publication
Hirata et al., Sci Rep. 2022 Jul 11;12:11757.
doi: 10.1038/s41598-022-15038-4
Researchers
平田 祥人 教授(筑波大学 システム情報系)
以下のフォームからお問い合わせください